ciencia, cáncer, "proteínas oscuras", genoma humano
 |
| Ilustración: Fabio Buonocore |
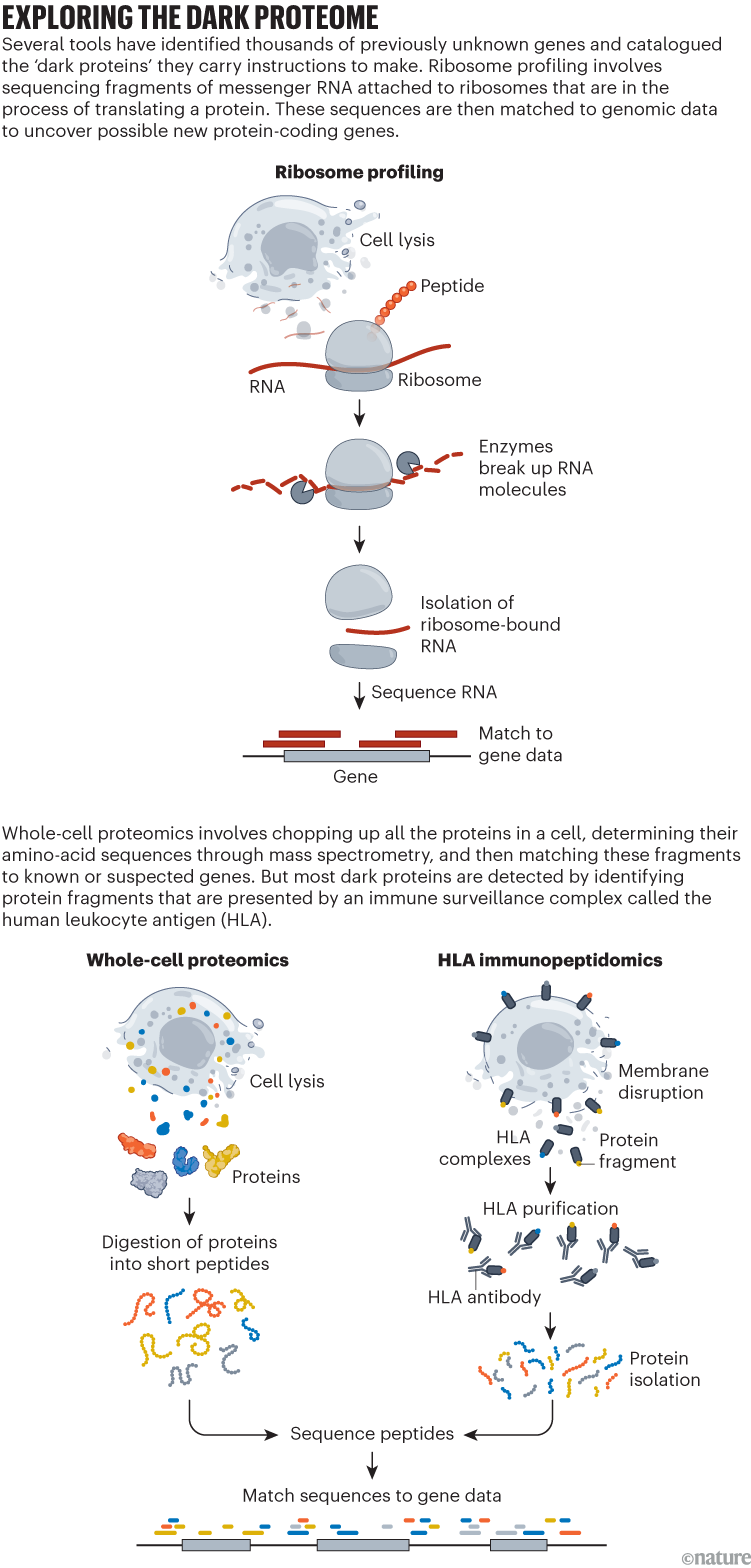
El genoma humano codifica miles de proteínas diminutas que antes se pasaban por alto. Ahora se está investigando para descubrir qué hacen.
30 enero 2025.- En 2009, Jonathan Weissman buscaba una nueva forma de espiar lo que sucede dentro de una célula . En particular, el biólogo celular molecular quería saber qué proteínas se producían en un momento determinado. Por eso, su laboratorio ideó una forma de medir directamente la producción de los ribosomas, las fábricas de proteínas de la célula.
El método, desarrollado con el entonces posdoctorado Nicholas Ingolia, que ahora está en la Universidad de California, Berkeley, implica recolectar todos los ribosomas de una célula y secuenciar las cadenas individuales de ARN mensajero que están unidas a ellos. Los investigadores esperaban que esta herramienta, llamada perfil de ribosomas, proporcionara un recuento preciso de todas las proteínas que produce una célula y sus cantidades relativas.
Pero, cuando Weissman y otros comenzaron a probar el método, se encontraron con una enorme sorpresa. Los ribosomas no sólo estaban produciendo a toda prisa proteínas codificadas por genes conocidos en el genoma de una célula, sino que también parecían estar creando miles y miles de "proteínas oscuras" que corresponden a partes del genoma que no se creía que produjeran proteínas 1 . "Ese fue el momento de revelación para nosotros", dice Weissman, que trabaja en el Instituto Whitehead en Cambridge, Massachusetts. Pronto, su laboratorio y otros descubrieron eventos de traducción inesperados en casi todos los organismos que examinaron.
Quince años después, los científicos todavía se preguntan qué hacer con estas proteínas.
Las proteínas oscuras tienden a ser cortas, a menudo de apenas unas pocas docenas de aminoácidos o menos. Y muchas son desconocidas, no tienen parientes cercanos en los genomas de otros organismos. Los estudios sugieren que algunas podrían tener funciones esenciales en la célula y podrían influir en la salud humana. Parecen ser abundantes en algunos tipos de cáncer, y varias empresas esperan desarrollar tratamientos que se dirijan a las proteínas oscuras. Pero en el caso de muchas de estas entidades misteriosas, la evidencia de que estén haciendo algo importante, o incluso de si sobreviven durante mucho tiempo en la célula, es equívoca.
El problema, dice Marie Brunet, que estudia proteómica en la Universidad de Sherbrooke (Canadá), es que los científicos no saben realmente qué es lo que les puede faltar. “Si falta una proteína en el repositorio, ni siquiera la están buscando”, afirma.
El enigma del recuento genético
Brunet forma parte de un esfuerzo global para documentar todas las proteínas oscuras codificadas por el genoma humano (véase "Explorando el proteoma oscuro"). El objetivo es atraer la atención de los investigadores hacia esta materia oscura, para que puedan empezar a averiguar qué hacen las proteínas, molécula por molécula.
Fuente: JR Prensner et al . Mol . Cell Prot . 22 , 100631 (2023).
Las perspectivas son apasionantes, dice Sebastiaan van Heesch, biólogo de sistemas del Centro Princesa Máxima de Oncología Pediátrica en Utrecht, Países Bajos, que también forma parte del esfuerzo: "Definitivamente hay nueva biología allí".
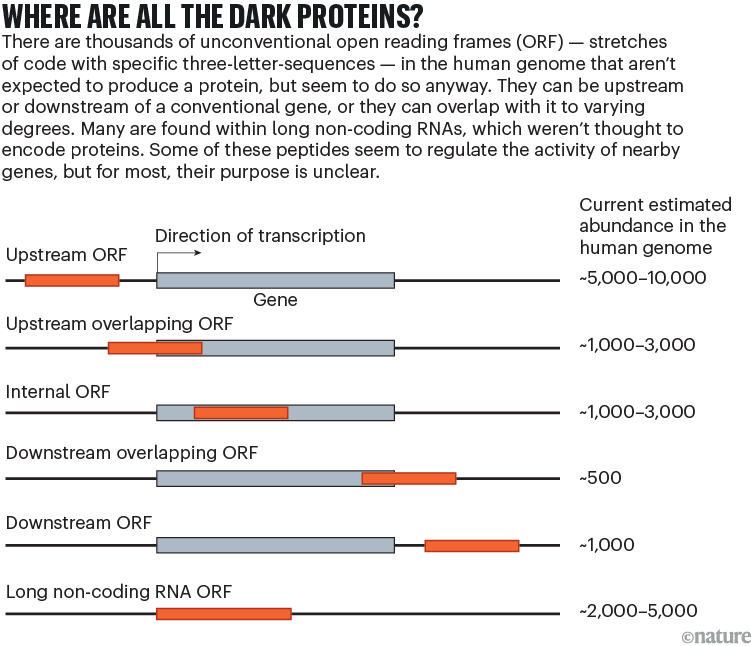
Antes de las primeras publicaciones del genoma humano a principios de la década de 2000, los investigadores examinaban frenéticamente los datos de secuencias emergentes tratando de estimar el número de genes codificadores de proteínas. Por lo general, buscaban lo que se conoce como marcos de lectura abiertos (ORF), tramos de código con secuencias específicas de tres letras, o codones, que pueden contener instrucciones para fabricar una proteína. Los genómicos buscaban más pistas, como evidencia de que una secuencia se conserva entre otros organismos y tiene una longitud razonable, todo lo cual son indicaciones de que la proteína resultante podría tener una función en las células.
Muchos ORFs que no cumplían estos criterios fueron ignorados o simplemente pasados por alto mientras los biólogos refinaban sus estimaciones. Los consorcios que mantienen listas de genes predichos, como el proyecto GENCODE, actualmente enumeran poco menos de 20.000 genes codificadores de proteínas. Alrededor del 90% de ellos han sido confirmados a través de otros esfuerzos, para producir proteínas correspondientes (los genes individuales pueden codificar múltiples proteínas diferentes, al incluir u omitir tramos de código llamados exones). GENCODE y otros proyectos realizan ajustes periódicos a sus listas a medida que surgen datos.
Pero, según John Prensner, biólogo oncológico de la Facultad de Medicina de la Universidad de Michigan en Ann Arbor, el recuento es incompleto. La idea de que los investigadores podían elaborar una lista completa de genes codificadores de proteínas ya en 2001 era un error muy extendido. “Los líderes del Proyecto Genoma Humano siempre supieron que apenas estaban iniciando una conversación”, afirma Prensner.
La capacidad de medir directamente la producción de los ribosomas provocó una explosión de interés en los ORFs pasados por alto y su potencial para codificar proteínas funcionales. En una correspondencia de 2022 con Nature Biotechnology , un equipo codirigido por Prensner, van Heesch y otros reunió una lista de más de 7000 de estos ORFs "no canónicos", que generalmente no cumplen los requisitos para ser considerados genes codificadores de proteínas y, por lo tanto, se han omitido de las bases de datos 2 . (Ese es un límite inferior, dice van Heesch; otros estudios han identificado decenas de miles de posibles proteínas oscuras codificadas por el genoma humano).
La mayoría de los ORF no convencionales que los científicos enumeraron se encuentran cerca de genes canónicos codificadores de proteínas o se superponen a ellos (véase "¿Dónde están todas las proteínas oscuras?"). Alrededor de un tercio de ellos se encontraban en secuencias llamadas ARN largos no codificantes, que, como sugiere el nombre, no se esperaba que codificaran proteínas, pero se pensaba que tenían funciones reguladoras.
Fuente: JR Prensner et al. Mol. Cell Prot . 22 , 100631 (2023)
Pero el hecho de que un ORF se traduzca en una proteína no significa que las proteínas sean estables o tengan funciones importantes en una célula. La traducción de algunos ORF no canónicos, que se lleva a cabo por los ribosomas, podría ser una forma de que las células controlen la actividad de un gen cercano, por ejemplo, obstruyendo la maquinaria ribosómica con productos que se degradarán rápidamente, dicen Prensner y otros. Este tipo de control se produce en ciertos ORF que aparecen antes de una secuencia codificante de proteínas.
En un estudio previo a la publicación de 2022, Prensner, van Heesch y un consorcio ampliado de especialistas en genómica y proteómica analizaron cientos de conjuntos de datos de proteómica (que comprenden miles de millones de puntos de datos) y hallazgos de estudios que utilizaron espectrometría de masas y otros enfoques para identificar el contenido proteico de las células 3 . Los investigadores encontraron fragmentos de proteínas correspondientes a más de 1.700 de los ORF no canónicos que habían identificado en 2022. Para 15 de ellos, argumentaron los investigadores, la evidencia era lo suficientemente sólida como para justificar la adición de las proteínas y sus genes correspondientes a los recuentos oficiales de genes codificadores de proteínas.
Pero, en el caso de la mayoría de los ORF no canónicos, no hay pruebas claras de que puedan producir una proteína. Parte del problema es el pequeño tamaño de las posibles proteínas (los investigadores las llaman microproteínas porque tienden a ser mucho más cortas que 100 aminoácidos; en promedio, las proteínas humanas contienen varios cientos de aminoácidos y muchas son mucho más largas). Su corta longitud dificulta la búsqueda de fragmentos coincidentes, que se crean en experimentos que separan las proteínas e identifican los fragmentos resultantes por su masa. Las muestras de células sobrerrepresentarán los fragmentos de proteínas más largas, dice van Heesch, en particular si las microproteínas son menos abundantes.
Cuando los científicos han utilizado la herramienta de inteligencia artificial llamada AlphaFold para predecir la estructura de las proteínas oscuras, las moléculas a menudo se parecen poco a las proteínas auténticas bien plegadas. Pero, dice Presner, "hay ejemplos claros que se parecen mucho a las proteínas canónicas y simplemente se pasaron por alto". GENCODE y otras organizaciones que administran repositorios han comenzado a agregar estas proteínas olvidadas a sus listas.
Jonathan Mudge, que trabaja en el proyecto GENCODE en el Instituto Europeo de Bioinformática del Laboratorio Europeo de Biología Molecular en Hinxton, Reino Unido, y es coautor de la preimpresión, dice que alrededor de 50 secuencias identificadas a través del perfil de ribosomas se han incluido en su lista de genes codificadores de proteínas humanas.
Pero se están moviendo con cuidado, añade. No son sólo los biólogos de laboratorio los que confían en esfuerzos como GENCODE para dar sentido a los experimentos. Los médicos también confían en esas bases de datos, y añadir una serie de genes codificadores de proteínas sospechosos podría complicar los esfuerzos para identificar variantes dañinas encontradas en los genomas de los pacientes, dice Mudge. "No somos escépticos. Sólo somos cautelosos", dice.
Ediciones para mayor claridad
En la misma época en que los investigadores empezaron a poner de relieve el potencial de los genomas para codificar proteínas oscuras, se produjo otro avance que permitió estudiar sistemáticamente sus efectos en las células: la edición genética CRISPR-Cas9. “De repente, pudimos extraer quirúrgicamente la secuencia codificante de estas proteínas no canónicas y preguntarnos si eran importantes para el funcionamiento de la célula”, afirma Weissman.
En un artículo de Science de 2020 , el equipo de Weissman demostró precisamente eso. Los investigadores utilizaron la edición genética CRISPR para interrumpir miles de ORF no canónicos, impidiendo que se tradujeran en proteínas en células pluripotentes inducidas humanas, así como en una línea de células cancerosas 4 . En cientos de casos, la edición CRISPR provocó un defecto de crecimiento en las células. "Muchas microproteínas eran realmente importantes para las células", dice Weissman.
Con más experimentos, pudieron determinar por qué. En algunos casos, las proteínas codificadas por un ORF no canónico interactuaban con una proteína codificada en la misma cadena de ARNm. Esto recuerda la forma en que los genes bacterianos corregulados tienden a residir uno al lado del otro en unidades llamadas operones, dice Weissman. Las proteínas oscuras funcionales que su equipo identificó asumieron diversas funciones en las células: una parecía estar involucrada en el ciclo celular, otra en la fisiología mitocondrial.
Las células cancerosas podrían ser especialmente ricas en proteínas oscuras. Prensner, que también es neurooncólogo pediátrico, está estudiando la posibilidad de que los ORF no canónicos presentes en todos los genomas humanos puedan estar desregulados en algunos cánceres, lo que podría contribuir a los diferentes resultados del tratamiento que él y otros médicos oncólogos ven en sus pacientes. "Estamos planteando la pregunta central de por qué los cánceres producen estas cosas", dice.
En experimentos similares a los de Weissman, un equipo dirigido por Prensner descubrió que aproximadamente el 10% de más de 500 ORFs no canónicos que inactivaron con CRISPR causaron defectos de crecimiento en varios tipos de células cancerosas humanas 5 . Prensner y sus colegas identificaron una proteína oscura que se expresaba en niveles elevados en líneas celulares de cáncer de mama y que también parecía impulsar su crecimiento.
En un estudio publicado el año pasado, Prensner, van Heesch y sus colegas identificaron varias proteínas oscuras que contribuyen a los meduloblastomas, cánceres cerebrales pediátricos a veces fatales 6 . En un ejemplo, los investigadores demostraron que una proteína oscura, independiente de una proteína canónica codificada por un ORF adyacente, estaba impulsando el crecimiento de formas especialmente agresivas de meduloblastoma que portan una versión hiperactiva de un gen del cáncer llamado MYC .
Estos ejemplos son la punta del iceberg, dice van Heesch, y se necesita mucho más trabajo para entender cómo funcionan las proteínas oscuras tanto en células sanas como enfermas, lo que subraya la necesidad de llamar la atención de otros científicos sobre estas proteínas. “Estudiar una de ellas lleva de tres a cuatro años. Estamos presentando miles”, dice. “Queda mucha biología por descubrir”.
Sacando la basura
Sin embargo, separar el trigo de la paja en este montón de posibilidades es una tarea abrumadora. “Una interpretación es que muchas de estas especies de proteínas que estamos observando de esta manera no son estables o no son funcionales, y en cierto modo reflejan el montón de basura”, dice Prensner. “La célula crea una secuencia de aminoácidos, la coloca en el compactador de basura y la escupe por el otro extremo”.
En el otro extremo se encuentra un sistema de vigilancia inmunológica que en los seres humanos se denomina complejo de antígeno leucocitario humano (HLA), que presenta fragmentos de proteínas en la superficie de las células. Este sistema ayuda a entrenar a las células inmunitarias para que no ataquen los tejidos normales y a alertarlas sobre amenazas, como virus y células cancerosas. Casi todas las cadenas de proteínas que fabrica una célula, incluso aquellas que no tienen ninguna función, terminan en exposición de esta manera.
Una gran parte de las proteínas oscuras que se identificaron en los seres humanos se encontraron utilizando una técnica que identifica los péptidos presentados por el complejo HLA. El hecho de que los investigadores hayan visto pocos otros signos de estas proteínas sugiere que la gran mayoría de ellas son errores de traducción o cumplen funciones únicamente para regular la traducción, dice Xuebing Wu, un biólogo molecular de la Universidad de Columbia en la ciudad de Nueva York. Sospecha que muchas proteínas oscuras ingresan a los sistemas de reciclaje celular demasiado rápido para asumir funciones definidas.
En un artículo de Nature de 2023 , Wu y sus colegas descubrieron que muchas proteínas oscuras llevan una firma química que las envía directamente del ribosoma a la basura 7 .
Pero la investigación también descubrió un mecanismo que dirige una pequeña fracción de proteínas oscuras a la membrana celular, donde están protegidas de la degradación y tienen la oportunidad de desarrollar nuevas funciones. Y en una revisión publicada a principios de este año, el equipo de Wu identificó alrededor de 1.000 proteínas oscuras que sospechan que son funcionales. Las células, dice, necesitan encontrar un equilibrio entre eliminar las proteínas oscuras potencialmente problemáticas sin sofocar la innovación.
Aunque la mayoría de las proteínas oscuras no tienen una función claramente definida, los científicos afirman que su presencia podría ser importante. El hecho de que muchas de ellas se encuentren solo cuando el sistema inmunitario presenta fragmentos de ellas podría sugerir funciones en la inmunidad, afirma van Heesch. Los ribosomas son orgánulos que consumen mucha energía y el coste de producir estas proteínas es “bastante tremendo”, afirma. “¿Por qué una célula traduciría basura una y otra vez?”
Sea lo que sea lo que las proteínas oscuras hacen en las células, las compañías farmacéuticas están empezando a considerarlas objetivos terapéuticos prometedores . Varias empresas de biotecnología, entre ellas Enara Bio en Oxford, Reino Unido, y Mnemo Therapeutics en París, están desarrollando terapias de células inmunes que se dirigen a los "antígenos oscuros" producidos por las células cancerosas y presentados por el complejo HLA. Un ensayo clínico que comenzará este año en China será uno de los primeros en apuntar al proteoma oscuro. El ensayo probará si una vacuna que alerta a las células inmunes sobre una proteína oscura producida por algunos cánceres de mama puede mejorar los resultados del tratamiento, después de que se observaran resultados prometedores en ratones 9 .
ProFound Therapeutics en Cambridge, Massachusetts, que Prensner asesora, está trabajando con el gigante farmacéutico Pfizer para apuntar al proteoma oscuro en busca de tratamientos para la obesidad.
Pero los científicos también están entusiasmados con la biología. Algunos especulan que las proteínas oscuras podrían representar un espacio de innovación genética en la evolución . “Quizás estemos presenciando el nacimiento de una proteína”, dice Weissman.
En un estudio de 2024 en Nature Cardiovascular Research , van Heesch y sus colegas identificaron un grupo de proteínas oscuras que parecen haber surgido en primates y en varios linajes de primates, incluidos los humanos, y algunas de ellas contribuyen a la función cardíaca 10 . Pero las innovaciones pueden tener un costo. Estas proteínas oscuras, más que los genes conservados, tienden a expresarse incorrectamente en células de personas con enfermedades cardíacas. Las proteínas oscuras que han adquirido funciones en el desarrollo normal pueden descontrolarse y causar cánceres, especialmente los que se observan en niños, agrega Prensner.
Los científicos que están arrojando luz sobre el proteoma oscuro afirman que no habrá una explicación única para lo que hacen estas moléculas en las células, y que las respuestas serán tan confusas y complicadas como el resto de la biología. Pero, más que nada, Prensner, van Heesch y otros científicos con ideas afines simplemente quieren que los biólogos presten atención a las proteínas y se preocupen menos por cómo categorizarlas.
Fuente: Nature
Referencias
1. Ingolia, N. et al. Science 324, 218–223 (2009). Artículo
2. Mudge, J. M. et al. Nature Biotechnol. 40, 994–999 (2022). Artículo
3. Deutsch, E. W. et al. Preprint at bioRxiv https://doi.org/10.1101/2024.09.09.612016 (2024).
4. Chen, J. et al. Science 367, 1140–1146 (2020). Artículo
5. Prensner, J. R. et al. Nature Biotechnol. 39, 697–704 (2021). Artículo
6. Hofman, D. A. et al. Mol. Cell 84, 261–276 (2024). Artículo
7. Kesner, J. S. et al. Nature 617, 395–402 (2023). Artículo
8. Azam, S., Yang, F. & Wu, X. Trends Genet. https://doi.org/10.1016/j.tig.2024.12.001 (2025). Artículo
9. Huang, D. et al. Nature 625, 593–602 (2024). Artículo
10. Ruiz-Orera, J. et al. Nature Cardiovasc. Res. 3, 1217–1235 (2024). Artículo

































COMENTARIOS